AG Schwamborn – Bildgebende Massenspektrometrie
Mitglieder:
PD Dr. med. Dr. nat. med. Kristina Schwamborn
Dr. rer. nat. Juliana Goncalves
Dr. med. Christine Bollwein
Um den zunehmenden Ansprüchen in Bezug auf Menge des zur Verfügung stehenden Gewebes, Zeitraum bis zur Diagnosestellung und Kosten gerecht zu werden, sind neue und innovative Methoden notwendig, die in Forschung und zukünftig auch in der pathologischen Routinediagnostik die Tumorsubtypisierung und Patienten-stratifizierung im Hinblick auf Therapieoptionen ermöglichen und erleichtern. Hierzu bietet sich die Analyse des Proteoms, d.h. des gesamtes Proteinäquivalents eines Genoms/ die Gesamtheit aller in einer Zelle oder einem Gewebe exprimierten Proteine an. Proteine steuern die Funktionen auf suprazellulärer, zellulärer und subzellulärer Ebene und stellen somit ein exaktes und sensitives Abbild zellulärer Vitalitätsparameter dar. So können sie als sensitive Sonde für Veränderungen im Gesundheitszustand der Zelle verwendet werden.
Um eine differentielle Charakterisierung des Proteoms zu ermöglichen, wurden im Laufe der letzten Jahrzehnte verschiedene Methoden entwickelt. Eine der wichtigsten dieser Methoden stellt die Massenspektrometrie (MS) dar. Für die klassischen proteomischen Methoden ist es nötig, dass vor der eigentlichen proteomischen Analyse Gewebe oder Zellen lysiert werden müssen. Daher gehen Informationen, die aus den Lagebeziehungen verschiedener Zelltypen zueinander resultieren können, verloren. Zusätzlich sollte der Zelltyp von Interesse für eine möglichst genaue und unverfälschte Analyse durch (Mikro-)Dissektion (manuell oder lasergestützt) isoliert werden. Hierin liegt einer der Vorteile der sogenannten bildgebenden Massenspektrometrie (Mass Spectrometry Imaging – MSI oder Imaging Mass Spectrometry – IMS). Erstmalig 1997 durch Richard M. Caprioli beschrieben, können mit ihr Massenspektren ortsaufgelöst von einem intakten Gewebeschnitt erzeugt werden. So kann auf elegante Weise die (Mikro-) Dissektion umgangen werden. Auch kleine Zellgruppen (und mit den neuesten Massenspektrometern sogar Einzelzellen) können vor einem komplexen Gewebehintergrund direkt evaluiert werden, ein Szenario, welches für jegliche andere proteomische Analysen (bisher) unrealistisch ist. Die Methode eignet sich im Besonderen für das Aufdecken neuer Biomarker, da Ziel-spezifische Reagenzien wie z.B. Antikörper sind nicht von Nöten sind. Die relative Menge von hunderten Molekülen (Proteine, (trytische) Peptide, Lipide, Glykane, Metabolite und Pharmazeutika) sowie deren Verteilung im Gewebe kann erfasst werden und mit klassischen morphometrischen, histochemischen und immunhistochemischen Analysen direkt korreliert werden. So erhält man hunderte von Molekülbildern, die wie in Analogie zu den verschiedenen Farbkanäle eines digitalen Bildes jeweils einem detektierten Molekül entsprechen (Abbildung 1).
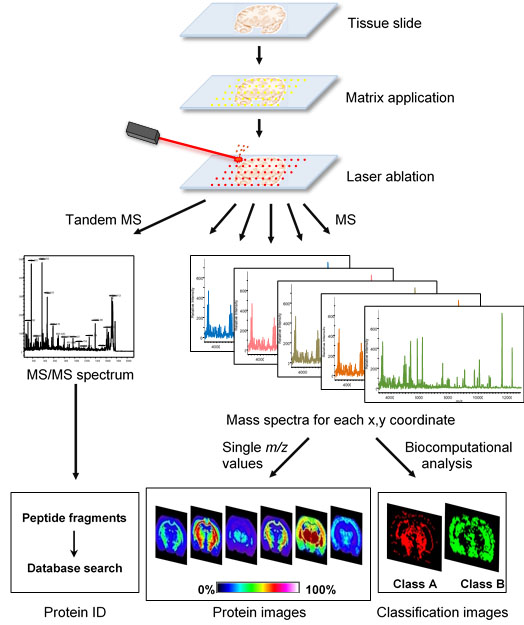
Unsere Arbeitsgruppe beschäftigt sich insbesondere mit der Anwendung der bildgebenden Massenspektrometrie in der Diagnostik, Charakterisierung und Subtypisierung verschiedener Tumoren. Durch die Kombination von Annotation und statistischer Datenanalyse werden Klassifizierungsalgorithmen erzeugt, die z.B. eine Unterscheidung verschiedener Tumoren voneinander erlauben.
Kollaborationen:
- Prof. Ron Heeren, M4i - Maastricht Multimodal Molecular Institute, Maastricht University, Niederlande
- Prof. Richard M. Caprioli, Mass Spectrometry Research Center Vanderbilt University School of Medicine, Nashville, TN, USA
- Prof. Peggi M. Angel, Cell and Molecular Pharmacology and Experimental Therapeutics, College of Medicine, Medical University of South Carolina, Charleston, SC, USA
- Dr. Rita Casadonte, Proteopath, Tier
- PD. Dr. Mark Kriegsmann, Institut für Pathologie, Universitätsklinikum Heidelberg
- CEP
- INTEGRATE-TN (Deutsche Krebshilfe)
Wir suchen für unser Team baldmöglichst eine/n Student/in. Mehr Infos...
Ausgewählte Publikationen:
Gonçalves JPL, Bollwein C, Noske A, Jacob A, Jank P, Loibl S, Nekljudova V, Fasching PA, Karn T, Marmé F, Müller V, Schem C, Sinn BV, Stickeler E, van Mackelenbergh M, Schmitt WD, Denkert C, Weichert W, Schwamborn K. Characterization of Hormone Receptor and HER2 Status in Breast Cancer Using Mass Spectrometry Imaging. Int J Mol Sci 2023, Vol 24, Page 2860. 2023;24(3):2860. doi:10.3390/IJMS24032860. PMID: 36769215
Gonçalves JPL, Bollwein C, Schlitter AM, Kriegsmann M, Jacob A, Weichert W, Schwamborn K. MALDI-MSI: A Powerful Approach to Understand Primary Pancreatic Ductal Adenocarcinoma and Metastases. Molecules. 2022;27(15):1-15. doi:10.3390/molecules27154811. PMID: 35956764
Gonçalves JPL, Bollwein C, Schwamborn K. Mass Spectrometry Imaging Spatial Tissue Analysis toward Personalized Medicine. Life 2022, Vol 12, Page 1037. 2022;12(7):1037. doi:10.3390/LIFE12071037. PMID: 35888125
Deininger S-O, Bollwein C, Casadonte R, Wandernoth P, Gonçalves, JPL, Kriegsmann K, Kriegsmann M, Boskamp T, Kriegsmann J, Weichert W, Schirmacher P, Ly A, Schwamborn K. Multicenter Evaluation of Tissue Classification by Matrix-Assisted Laser Desorption/Ionization Mass Spectrometry Imaging. Anal Chem, 2022, 94, 23, 8194–8201. doi:10.1021/ACS.ANALCHEM.2C00097. PMID: 35658398
Bollwein C, Gonҫalves JPL, Utpatel K, Weichert W, Schwamborn K. MALDI Mass Spectrometry Imaging for the Distinction of Adenocarcinomas of the Pancreas and Biliary Tree. Molecules. 2022;27(11):3464. doi:10.3390/MOLECULES27113464. PMID: 35684402
Gonçalves JPL, Bollwein C, Schlitter AM, Martin B, Märkl B, Utpatel K, Weichert W, Schwamborn K. The Impact of Histological Annotations for Accurate Tissue Classification Using Mass Spectrometry Imaging. Metabolites. 2021 Oct 30;11(11):752. doi: 10.3390/metabo11110752. PMID: 34822410
Lopes Gonçalves JP, Bollwein C, Weichert W, Schwamborn K. Implementation of Mass Spectrometry Imaging in Pathology: Advances and Challenges. Clin Lab Med. 2021 Jun;41(2):173-184. doi: 10.1016/j.cll.2021.03.001. PMID: 34020758 Review.
Martin B, Gonçalves JPL, Bollwein C, Sommer F, Schenkirsch G, Jacob A, Seibert A, Weichert W, Märkl B, Schwamborn K. A Mass Spectrometry Imaging Based Approach for Prognosis Prediction in UICC Stage I/II Colon Cancer. Cancers (Basel). 2021 Oct 26;13(21):5371. doi: 10.3390/cancers13215371. PMID: 34771536
Kriegsmann M, Zgorzelski C, Casadonte R, Schwamborn K, Muley T, Winter H, Eichhorn M, Eichhorn F, Warth A, Deininger SO, Christopoulos P, Thomas M, Longerich T, Stenzinger A, Weichert W, Müller-Tidow C, Kriegsmann J, Schirmacher P, Kriegsmann K. Mass Spectrometry Imaging for Reliable and Fast Classification of Non-Small Cell Lung Cancer Subtypes. Cancers (Basel). 2020 Sep 21;12(9):E2704. PMID: 32967325
Schwamborn K, Weirich G, Steiger K, Zimmermann G, Schmidmayr M, Weichert W, Caprioli RM. Discerning the Primary Carcinoma in Malignant Peritoneal and Pleural Effusions Using Imaging Mass Spectrometry-A Feasibility Study. Proteomics Clin Appl. 2019 Jan;13(1):e1800064. PMID: 30311431
Angel PM, Schwamborn K, Comte-Walters S, Clift CL, Ball LE, Mehta AS, Drake RR. Extracellular Matrix Imaging of Breast Tissue Pathologies by MALDI-Imaging Mass Spectrometry. Proteomics Clin Appl. 2019 Jan;13(1):e1700152. PMID: 30251340
Ly A, Longuespée R, Casadonte R, Wandernoth P, Schwamborn K, Bollwein C, Marsching C, Kriegsmann K, Hopf C, Weichert W, Kriegsmann J, Schirmacher P, Kriegsmann M, Deininger SO. Site-to-Site Reproducibility and Spatial Resolution in MALDI-MSI of Peptides from Formalin-Fixed Paraffin-Embedded Samples. Proteomics Clin Appl. 2019 Jan;13(1):e1800029. doi: 10.1002/prca.201800029. PMID: 30408343
Leung F, Eberlin LS, Schwamborn K, Heeren RMA, Winograd N, Cooks RG. Mass Spectrometry-Based Tissue Imaging: The Next Frontier in Clinical Diagnostics? Clin Chem. 2019 Apr;65(4):510-513. PMID: 30737204
Spraggins JM, Schwamborn K, Heeren RMA, Eberlin LS. The Importance of Clinical Tissue Imaging. Clin Mass Spectrom. 2019 Apr;12:47-49. PMID: 32483555
Porta Siegel T, Hamm G, Bunch J, Cappell J, Fletcher JS, Schwamborn K. Mass Spectrometry Imaging and Integration with Other Imaging Modalities for Greater Molecular Understanding of Biological Tissues. Mol Imaging Biol. 2018 Dec;20(6):888-901. PMID: 30167993
Schwamborn K, Kriegsmann M, Weichert W. MALDI imaging mass spectrometry – From bench to bedside. Biochim Biophys Acta. 2017 Jul;1865(7):776-783. PMID: 27810414
Schwamborn K. The Importance of Histology and Pathology in Mass Spectrometry Imaging. Adv Cancer Res. 2017;134:1-26. PMID: 28110647
Kriegsmann M, Casadonte R, Kriegsmann J, Dienemann H, Schirmacher P, Kobarg JH, Schwamborn K, Stenzinger A, Warth A, Weichert W. Reliable entity subtyping in Non-small cell Lung Cancer by MALDI Imaging Mass Spectrometry on Formalin-fixed Paraffin-embedded Tissue Specimens. Mol Cell Proteomics. 2016 Oct;15(10):3081-3089. PMID: 27473201
Schwamborn K, Caprioli RM. Molecular imaging by mass spectrometry — looking beyond classical histology. Nat Rev Cancer. 2010 Sep;10(9):639-46. PMID: 20720571
Schwamborn K, Caprioli RM. MALDI imaging mass spectrometry – painting molecular pictures. Mol Oncol. 2010 Dec; 4(6):529-38. PMID: 20965799
Schwamborn K. Imaging mass spectrometry in biomarker discovery and validation. J Proteomics. 2012 Aug 30;75(16):4990-8. PMID: 22749859